流れ 2004年12月号 目次
― 特集:ナノ・マイクロスケールの熱流動 ―
| リンク一覧にもどる | |
DNAナノデバイス創製における流動ダイナミクス
 東北大学学際科学 国際高等研究センター 科学技術振興機構 川野聡恭 |
概要
半導体製造等に用いられている超微細加工技術は著しい発展を遂げてきたが,フォトリソグラフィ-およびプラズマエッチングに基づく加工線幅のさらなる微細化には,光の波長に起因した限界が近づいている.DNAは塩基対のπスタッキングを介した電気伝導性を持つので,線幅約2nmの導線として利用可能であり,また,自己組織化機能や分子認識機能を有し,生体との親和性も高い.guanine-cytosine DNAがp型半導体,adenine-thymine DNAがn型半導体になることも実験的に示唆されている.以上のことから,DNAによる電子デバイスは革命的新技術として期待されている.また,DNAの熱変性機能やB型からZ型への構造転換機能を有効に利用し,これを駆動源としたDNAナノ機械の創製も欧米では活発に研究開発がなされている. 本研究グループでは,その自己組織化機能を有効に利用したDNAデバイス製造法について,基礎設計指針の提案を目標とした研究開発を理論ならびに実験の両面から行っている.DNAの機能発現やDNAナノ機械の製造は必ず水溶液中で行われることから,水分子と生体高分子の相互干渉問題を特に重視し,分子流体工学を基盤として,量子化学,生命科学,電子工学および材料科学を先端融合化した学際研究「バイオ・ナノ流動ダイナミクス」の学理構築を目指す. 本稿では,poly(dA)・poly(dT) 人工DNAの固体基板近傍における自己組織化流動現象を取り上げ,これに関する実験およびシミュレーション技術について簡単に解説する.
固体基板近傍におけるDNAの自己組織化流動現象
原子間力顕微鏡による観察
本実験で使用するDNAサンプルは,長さが17nm,直径が2nmの50塩基対poly (dA)・poly (dT) 人工DNA 2本鎖である.超純水を用いてDNA滴下用溶液を作製し,これをマイカ(雲母)新規面上に滴下して自然乾燥させたものを観察試料とした.試料の観察は原子間力顕微鏡 (AFM) のタッピングモードによって行われた.
図1は,DNAがマイカ基板近傍で自己組織化流動現象を呈し,基板上にネットワーク構造として固着した結果のAFM画像である.ネットワーク構造は,濃度80μg/mlのDNA溶液を3μl滴下して作成された試料から得られたものである.図1(a) は三次元像であり,網目の大きさは不規則であるものの,比較的高さのそろったネットワークがほぼ均一に形成されていることがわかる.ボックスカウンティング法により得られた二次元および三次元のフラクタル次元はそれぞれ1.63および2.81であった.また,図1(b) に示したネットワーク二次元像の2点ABにおける断面図を図1(c) に示す.AB間のDNAネットワークは高さが約1.2-2.0nm,太さが約52.6-84.1nmであり,以上のことから,このネットワーク構造は,一本の高さがおおむねDNAの直径と同じで,数十本のDNAが基板と水平にバンドルしていることが示唆された.将来,基板‐水分子‐DNA分子の相互干渉を精密に制御することにより,このネットワーク形成機能を配線技術に応用することが大いに期待されている.
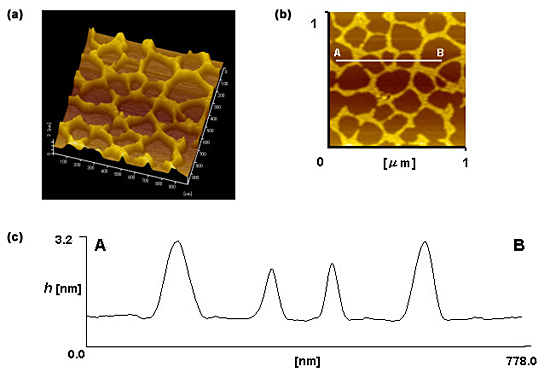
図1 マイカ基板に固着したpoly (dA)・poly (dT) DNAネットワークのAFMによる観察結果
さらに本実験では,ネットワークの同一部位をタッピングモードとノンコンタクトモードで観察し,得られた2つの像を比較検討した.図2(a) はノンコンタクトモード,(b) はタッピングモードで観察されたマイカ上のDNAネットワークである.観察試料の作成には濃度20µg/mlのDNA溶液を3µl使用した.選択された線分ABにおけるDNAネットワーク断面図を二次元像の右に示す.両モードともDNAネットワーク性状をよく捉えているが,ノンコンタクトモードでは信号にノイズが多く見られる.また,カンチレバーの接触による生体試料の損傷はほとんど見られないことから,タッピングモード観察の有効性が示されたと言える.ノンコンタクトモード測定で得られたDNAネットワークの高さは約0.8-0.9nm,太さは約24.6-42.6nmであり,タッピングモード測定で得られた高さは約1.0-1.3nm,太さは約42.5-58.2nmであった.測定値には比較的大きな差が見られるが,DNAネットワークの基本構造(高さ方向にはDNA 1本が歪んで固着しており,横方向にはDNAが数十本バンドルしている)の定性的傾向は両モードとも正しく捉えている.
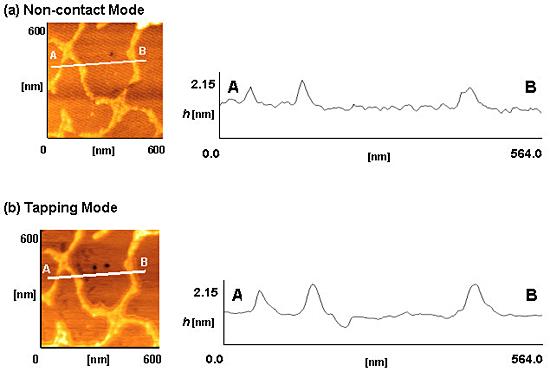
図2 ノンコンタクトモードとタッピングモードによるDNAネットワークの観察
コンピュータシミュレーション
本研究グループでは以下のステップを経て,基板上に配置されたpoly(dA)・poly(dT)DNA水溶液をコンピューター上で再現する.
1.Gaussian あるいはAmber (Nucgen) を用いて10塩基対poly(dA)・poly(dT)DNA断片の位置座標をpdb(プロテインデータバンク)形式で作成後,5'末端および3'末端を水素終端処理する.
2.リン酸基へのカウンターカチオンとしてNa+を付加し,poly(dA)・poly(dT)DNAを電気的に中性化する.
3.基板とpoly(dA)・poly(dT) DNAを含む直方体領域をH2O分子で満たす.
4.基板を含む各原子に量子化学計算によって得られるMulliken 電荷を与える.
次にシミュレーションの流れであるが,現実的な状態(平衡状態)へ少しでも早く近づけるため,プリプロセスとして系の緩和を行った後でMD(分子動力学)計算を実行する.すなわち,
1.DNAの基本形状が崩れないようにDNA構成原子を拘束したまま水分子とカウンターイオンのエネルギーを最小化することで系を緩和.
2.DNA,水分子,カウンターイオンの全エネルギーを最小化し,系全体を緩和.
3.DNAの基本形状が崩れないようにShake法により水素原子を含む結合を拘束し,温度を0Kから300Kに上昇させながら計算を実行する.
4.1-3を経て300Kにて計算ができるようになった状態を平衡状態0psとし,再びShake法により水素原子を含む結合を拘束し,全体の系をNTPアンサンブル(原子数・温度・圧力一定)にてMD計算を行う.ただし,遠距離力であるクーロン力の算出には Particle Mesh Ewald法を用いる.
著者らは分子動力学シミュレーションにおいて,著名な市販ソフトであるAmberの力場を完全に含み,生体高分子の大変形や高温条件にも対応した力場ポテンシャルを提案し,並列化MDシミュレーションにおいて,その有用性を確認している.すなわち,Amber Force Field (力場)ポテンシャルは次式で表される.
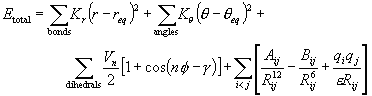
これは,原子間の結合,角度,ねじれ(二面角),ファンデルワールス力(クーロン力を含む)の項から成り, 2nd Generation Force Fieldとして広く知られている.しかしながら,右辺第1項および第2項では,r![]() reqあるいはθ
reqあるいはθ![]() θeqでなければ,ポテンシャルが急激に発散することが明白で,これは,計算結果であるはずの分子構造が,pdbにあるX線回折等による構造解析結果,すなわちreq およびθeqと大きく違わないような系でのみ有用である.現在まで著者らは,DNA等の生体高分子に関するMDシミュレーションコードの基幹部分を完成させ,とくに,DNAの複製過程やたんぱく質の機能発現には,その二次構造が非常に重要で,生体高分子の大変形も考慮しなければならず,前述のポテンシャルでは取り扱いが困難であることを示した.そこで,著者らは,結合項を次のようなモースポテンシャルに変換した独自のコードを作成した.
θeqでなければ,ポテンシャルが急激に発散することが明白で,これは,計算結果であるはずの分子構造が,pdbにあるX線回折等による構造解析結果,すなわちreq およびθeqと大きく違わないような系でのみ有用である.現在まで著者らは,DNA等の生体高分子に関するMDシミュレーションコードの基幹部分を完成させ,とくに,DNAの複製過程やたんぱく質の機能発現には,その二次構造が非常に重要で,生体高分子の大変形も考慮しなければならず,前述のポテンシャルでは取り扱いが困難であることを示した.そこで,著者らは,結合項を次のようなモースポテンシャルに変換した独自のコードを作成した.
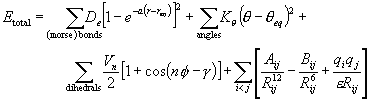
右辺第一項は,r![]() reqを仮定して線形近似すれば,AMBERコードにおける右辺第一項と一致するため,我々のモデルは従来のモデルを完全に含有し,しかも,大変形ダイナミクスの取り扱いが可能となるように拡張されたことになる.物質毎のパラメータも簡単な理論解析により,既存のデータベースを利用して決定することができる.
ここでは,水素終端したSiO2基板に固着する10塩基対poly(dA)・poly(dT)DNA の流動ダイナミクスシミュレーションを行った.t=900psまでの結果を図3示す.明瞭な可視化の為,H2O分子を無視している.時間が経過するにつれ,DNAの形状は変化し,5'末端と3'末端が基板に付着していく様子が再現されている.
ただし,より厳密に言えば,経験則に基づく力場ポテンシャルを用いている限り,分子の励起状態や振動など,量子力学的現象が支配的であるような物理・生命現象には不向きであることがわかる.θ方向への大変形問題も未解決であり,生体高分子の立体構造をある程度維持するために導入される拘束手法の開発も急務である.また,Mulliken電荷の設定による量子力学効果の取り込みが可能になっているが,pHや電荷の時空間変化が,生命機能発現・維持において本質的であるから,さらなる力場ポテンシャルの高精度化と的確な量子力学的効果との融合化が急務となっている.
reqを仮定して線形近似すれば,AMBERコードにおける右辺第一項と一致するため,我々のモデルは従来のモデルを完全に含有し,しかも,大変形ダイナミクスの取り扱いが可能となるように拡張されたことになる.物質毎のパラメータも簡単な理論解析により,既存のデータベースを利用して決定することができる.
ここでは,水素終端したSiO2基板に固着する10塩基対poly(dA)・poly(dT)DNA の流動ダイナミクスシミュレーションを行った.t=900psまでの結果を図3示す.明瞭な可視化の為,H2O分子を無視している.時間が経過するにつれ,DNAの形状は変化し,5'末端と3'末端が基板に付着していく様子が再現されている.
ただし,より厳密に言えば,経験則に基づく力場ポテンシャルを用いている限り,分子の励起状態や振動など,量子力学的現象が支配的であるような物理・生命現象には不向きであることがわかる.θ方向への大変形問題も未解決であり,生体高分子の立体構造をある程度維持するために導入される拘束手法の開発も急務である.また,Mulliken電荷の設定による量子力学効果の取り込みが可能になっているが,pHや電荷の時空間変化が,生命機能発現・維持において本質的であるから,さらなる力場ポテンシャルの高精度化と的確な量子力学的効果との融合化が急務となっている.
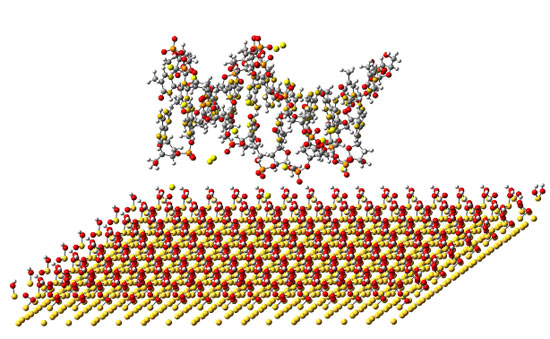
t=0ps
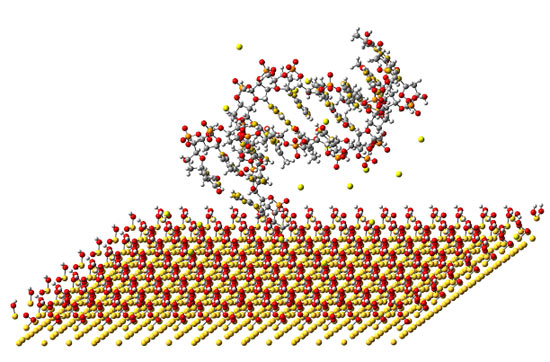
t=200ps
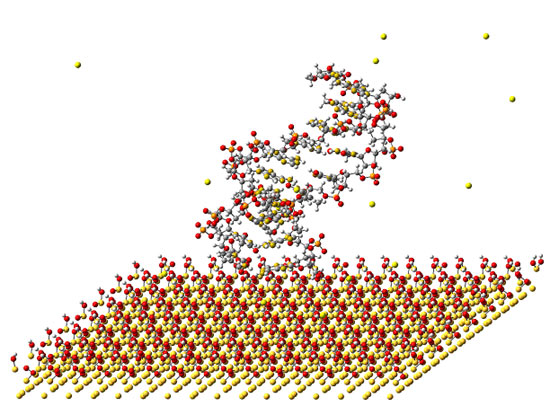
t=900ps
図3 SiO2基板に固着するpoly(dA)・poly(dT) DNA の流動ダイナミクスシミュレーション
文献
- Naoto SHIMIZU, Satoyuki KAWANO, and Masanori TACHIKAWA Electron Correlated and Density Functional Studies on Hydrogen-Bonded Proton Transfer in Adenine-Thymine Base Pair of DNA J. Molecular Structure, in press.
- M. KAMPP, Satoyuki KAWANO, P.J.P. ROCHE, J. RASCH, D.H. MADISON, H.R.J. WALTERS and C.T. WHELAN On the Observation of the Fine Structure Effect in Non-Relativistic (e, 2e) Processes Eur. Phys., D, Vol.29(2004), pp.17-19
- 川野聡恭, 志賀智行 生体高分子の大変形を考慮した拘束系MDシミュレーション手法の開発 シミュレーション, 第23巻, 第1号(2004), pp.42-47.
- 川野聡恭, 丸山洋平 電子衝突によるDNAらせん崩壊の量子力学的アプローチ シミュレーション, 第23巻, 第1号(2004), pp.36-41.
- Satoyuki KAWANO Fractal Dimension Analysis in Self-assembled Poly(dA)?Poly(dT) DNA Network on Mica Surface Proceedings of the Fourth International Symposium on Advanced Fluid Information and the First International Symposium on Transdisciplinary Fluid Integration, Sendai, Japan, (2004), pp.38-39.
- Youhei MARUYAMA, Masanori TACHIKAWA, Satoyuki KAWANO Ab Initio Study of DNA Double Strand Breaks by Hydroxyl Radical Proceedings of the Fourth International Symposium on Advanced Fluid Information and the First International Symposium on Transdisciplinary Fluid Integration, Sendai, Japan, (2004), pp.40-41.
- Satoyuki KAWANO, Tomoyuki SHIGA, Chikako TAKATOH, Tomoji KAWAI Molecular Dynamics Simulation in Nanowiring of Poly(dG)-Poly(dC) DNA on Highly Orientated Pyrolytic Graphite MICRO SYSTEM Technologies 2003, pp. 568-570, Franzis Verlag GmbH.
謝辞
本稿をまとめるにあたり,芳賀真弓氏(科学技術振興機構研究補助員),川原梢氏(東北大学研究支援者)および長井清香氏(東北大学研究支援者)のご助力を得た.ここに付記し謝意を表する.

